[Nota: si no sabes interpretar las hipótesis embebidas en el código que publico, que operan como enormes caveats, no hagas caso en absoluto a los resultados. He publicado esto para ver si otros que saben más que yo lo pulen y consiguen un modelo más razonable usándolo tal vez, ojalá, como núcleo.]
[Edición: He subido el código a GitHub.]
[El código de esta sección y los resultados contienen errores de bulto; consúltese el código de GitHub.]
Escribo casi al vuelo e inspirado por la idea de que:
- Los casos verificados de coronavirus no son fiables. Ni están todos los que son, ni son todos los que están.
- Los casos de defunciones son mucho más fiables: son menos y se les presta mucha atención.
La idea la he expresado públicamente en:
Problema estadístico: ¿cómo estimar el número de casos activos? Pensemos: los casos confirmados no son fiables. Los más fiables son los fallecidos. Tenemos una variable observada, fallecidos, "producto" de otra inobservada, "activos", como en un HMM (modelo oculto de Markov).
— Carlos Gil Bellosta (@gilbellosta) March 19, 2020
Y aquí van los resultados crudísimos de un modelo crudísimo y seguramente erróneo cuya convergencia ni he constrastado ni nada:
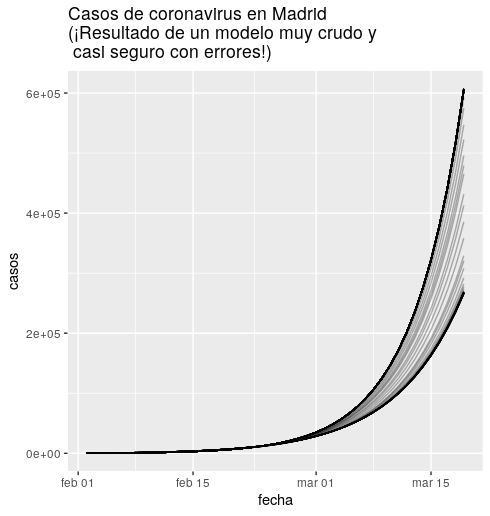
Y el código es:
data {
int<lower=0> N;
int<lower=0> dia0;
vector[N] dia;
vector[N] defs;
}
parameters {
real<lower = 0, upper = 1000> casos_0;
vector[N] contagios;
real<lower = 0, upper = 3> r0; //priori cutre sobre r0, infectados diarios que genera un caso
real<lower = 0, upper = .02> letalidad;
}
model {
vector[N] casos;
contagios[1] ~ normal(casos_0 * r0, 1);
casos[1] = casos_0 + contagios[1];
for (i in 2:N){
contagios[i] ~ normal(casos[i-1] * r0, 1);
casos[i] = casos[i-1] + contagios[i];
}
for (i in dia0:N) {
defs[i] ~ normal(letalidad * casos[i - 22], 1);
}
}y
library(rstan)
library(reshape2)
library(plyr)
library(ggplot2)
dat <- read.csv("https://raw.githubusercontent.com/datadista/datasets/master/COVID%2019/ccaa_covid19_fallecidos.csv")
dat <- melt(dat, id.vars = c("cod_ine", "CCAA"))
dat$fecha <- as.Date(dat$variable, format = "X%d.%m.%Y")
fecha_ini <- min(dat$fecha)
madrid <- dat[dat$CCAA == "Madrid",]
madrid$dia <- as.numeric(madrid$fecha - fecha_ini)
madrid <- madrid[, c("dia", "value")]
colnames(madrid) <- c("dia", "defs")
tmp <- data.frame(dia = -30:-1, defs = 0)
madrid <- rbind(tmp, madrid)
fit <- stan(file = "stan.stan",
data = list(N = nrow(madrid), dia0 = which(madrid$dia == 0), dia = madrid$dia, defs = madrid$defs),
iter = 10000, warmup = 2000,
chains = 1, thin = 10)
res <- as.data.frame(fit)
contagios <- extract(fit, pars = "contagios")$contagios
contagios <- data.frame(t(contagios))
contagios$fecha <- as.Date(madrid$dia, origin = fecha_ini)
contagios <- melt(contagios, id.vars = "fecha")
casos <- ddply(contagios, .(variable), transform, casos = cumsum(value))
ggplot(casos, aes(x = fecha, y = casos, group = variable)) +
geom_line(alpha = 0.3) +
xlab("fecha") + ylab("casos") +
ggtitle("Casos de coronavirus en Madrid\n(¡Resultado de un modelo muy crudo y\n casi seguro con errores!)")